Subiecte
rezumat
Picornavirusul slab studiat, paregovirusul 3 uman (HPeV3) provoacă sepsis neonatal fără terapii disponibile. Structura noastră de rezoluție 4.3-Å a HPeV3 pe cont propriu și rezoluția de 15 Å în complexe cu anticorpi monoclonali umani Fab demonstrează structura așteptată a capsidei picornavirusului cu trei caracteristici distincte. În primul rând, 25% din genomul ARN HPeV3 de la 60 de situri este foarte ordonat, confirmat de reconstrucția asimetrică, și interacționează cu regiunile conservate ale proteinelor capsidei VP1 și VP3. În al doilea rând, termenul VP0 N stabilizează suprafața interioară a capsidei, spre deosebire de alte picornavirusuri în care la ejecție, cum ar fi VP4, formează un canal de translocație ARN. În cele din urmă, buzunarul hidrofob al VP1, locul de legare al medicamentului antipicornaviral, pleconaril, este blocat și, prin urmare, nepotrivit pentru dezvoltarea antivirală. Luate împreună, aceste rezultate sugerează o direcție pentru dezvoltarea anticorpilor neutralizanți, a medicamentelor antivirale bazate pe interacțiunile ARN-proteină și disecția asamblării virusului pe baza nucleației ARN.
Introducere
Picornaviridae este o familie de viruși ARN monocatenari, cu simulare pozitivă, icosaedrică. Parechovirusul A este o specie din această familie cu 16 genotipuri și este în principal asociat cu infecții ușoare la om, în special la copii. Cu toate acestea, un agent patogen emergent, parevirusul 3 uman (HPeV3) poate provoca infecții grave în sistemul nervos central, cum ar fi meningita 1, și este o cauză principală a sepsisului neonatal 2. Nu există antivirale sau vaccinuri disponibile pentru combaterea infecției cu HPeV. Spre deosebire de multe alte picornavirusuri, HPeV-urile sunt slab caracterizate atât în ceea ce privește structura, cât și funcționalitatea, cu excepția HPeV1, unde este cunoscut receptorul 3. Diferențele mari de tropism prezentate de HPeV3 comparativ cu celelalte HPeV, fac esențială investigarea proprietăților structurale ale HPeV3 pentru o mai bună înțelegere a patogenezei sale și a unei posibile legări a receptorilor.
Folosim microscopia crioelectronică și reconstrucția imaginii pentru a analiza structura HPeV3 de la sine și în complex cu un anticorp monoclonal Fab uman. Structura virionului arată că medicamentele care leagă buzunarul VP1, cum ar fi pleconarilul, este puțin probabil să se lege de HPeV; că VP0 este o proteină importantă pentru stabilizarea suprafeței interioare a capsidei și, în cele din urmă, că ansamblul HPeV este probabil controlat de interacțiuni multiple ale genomului cu capsida, prin aminoacizi conservați în VP1 și VP3 și structuri stem și bucle în ARN. Izolăm și caracterizăm un anticorp monoclonal uman specific pentru HPeV3, care ar putea fi foarte util pentru a avansa diagnosticul virușilor și a studia interacțiunile virus-gazdă.
Rezultate si discutii
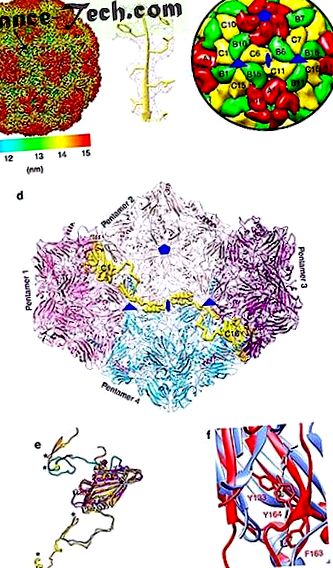
Structura HPeV3
Imagine la dimensiune completă
Structura ordonată a ARN-ului HPeV3
Imagine la dimensiune completă
Imagine la dimensiune completă
Buzunar hidrofob VP1
Medicamentul antiviral, pleconarilul și derivații săi blochează în general infecțiile cu enterovirus, cum ar fi EV-D68 (ref. 4). Aceste medicamente expulzează lipida („factorul de buzunar”) prezent în buzunarul hidrofob al β-butoiului VP1 și blochează învelișul de ARN prin stabilizarea capsidei 4. Canalul către buzunarul hidrofob din HPeV3 VP1 este blocat de trei lanțuri laterale mari conservate Y133, F163 și Y164 (Fig. 1f și 3) comparativ cu EV-D68 (ref. 4). Prin urmare, putem explica observațiile conform cărora pleconarilul nu funcționează în clinica 2 și putem prezice că astfel de factori de buzunar nu vor funcționa împotriva oricărui HPeV secvențiat până acum.
HPeV3 - Structură complexă Fab
Am izolat un anticorp monoclonal uman specific HPeV3, AT12-015, care s-a legat de izolatele HPeV3 152037, A308/99 și două izolate clinice din celulele infectate. Nu a neutralizat 152037 într-un test bazat pe liniile celulare Vero. Structura izolatului HPeV3 152037 a fost rezolvată în complex cu Fab AT12-015 (Tabelul suplimentar 1; Fig. 4a; Fig. Suplimentară 1). Moleculele Fab recunosc un epitop conformațional pe marginea butoiului (Fig. 4a). Contribuțiile provin din regiuni atât în VP3 (bucle hZβB, βChA, βEβF și βGβH), cât și în VP1 (βBβC, βCβD și βEβF, C) conservate în principal în HPeV3, ceea ce explică specificitatea anticorpului la acest genotip (Fig. 3) . Amprenta Fab se întinde pe terminalul VP1 C, unde multe alte HPeV conțin un motiv RGD pentru a se lega de receptorii lor de integrină (Fig. 3 și 4b) 3. Deoarece AT12-015 este specific pentru HPeV3 și nu s-a legat de niciunul dintre celelalte genotipuri HPeV testate, ar putea fi foarte util în avansarea diagnosticării virusului și studierea interacțiunilor virus-gazdă. O comparație suplimentară cu epitopii serurilor de pacienți neutralizanți ne-ar putea ajuta să înțelegem mecanismul de neutralizare la pacienți 13 .
( la ) Reprezentarea izosurfială a culorii radiale a complexului HPeV3-Fab AT12-015 la rezoluția de 15 Å prezentată la 1,5σ peste pragul mediu. Moleculele Fab (roșii) se leagă în jurul regiunii canionului. ( b ) Foaia de parcurs HPeV3. Amprenta Fab (contur roșu) se mapează la VP3 (verde) și VP1 (roșu).
Imagine la dimensiune completă
În general, această lucrare arată interacțiuni ARN-capsidă stabilizatoare multiple ca o nouă țintă pentru medicamentele cu parevirus. Dezvăluim stabilitatea neobișnuită a acestor capside la nivelul intra-pentamerului prin interacțiunile VP3 și la inter-pentamerul prin VP0 și propunem că mecanismul de acoperire a genomului HPeV va fi semnificativ diferit de alte picornavirusuri.
Metode
Purificarea și inactivarea virusului.
HPeV1-Harris și HPeV2-2008 au fost furnizate de Institutul Național Olandez pentru Sănătate Publică și Mediu, Bilthoven, Olanda. HPeV4-251176, HPeV5-552322, HPeV6-550389 și două izolate clinice HPeV3 au fost obținute de la Laboratorul de virologie clinică, Centrul Medical Academic, Amsterdam, Olanda. Pentru stocurile de virus HPeV1, HPeV2, HPeV3 A308/99 (tip cadou Hiroyuki Shimizu și Miyabe Ito) și HPeV4-6 au fost cultivate într-o linie celulară HT29 în mediul esențial minim Eagle cu acid L-glutamic (0, 2 ×), aminoacizi neesențiali (1x), streptomicină (0,1 μg ml -1), penicilină (0,1 μg ml -1) și 2% FBS inactivat la căldură (celulele HT29 au fost menținute în mediu conținând 8% FBS căldură inactivată). Aceste tulpini au fost utilizate în testele de legare și neutralizare a anticorpilor AT12-015.
Generarea de anticorpi și prepararea fragmentelor Fab.
Anticorpul monoclonal uman AT12-015 împotriva izolatului HPeV3 152037 a fost preparat din sânge uman 15. Pe scurt, memoria umană celulele CD27 + IgG + B au fost cultivate folosind metoda AIMSelect de la donatori sănătoși care s-au recuperat dintr-o infecție izolată 152037 cu HPeV3 confirmată cu 1 an mai devreme. Celulele unice au fost subclonate din culturi de celule B în care supernatantul a prezentat legarea de celule infectate cu HPeV3 prin imunofluorescență. ARN-ul a fost izolat din celulele B monoclonale pentru a recupera secvențele lanțului greu și ușor ale anticorpului. Secvențe unice au fost utilizate pentru a genera proteine recombinante în 293 celule T. Anticorpii IgG1 au fost purificați în continuare folosind coloane de proteină A HiTrap pe un instrument ÄKTA (GE). Fragmentele Fab AT12-015 au fost produse folosind un kit de microprep Pierce Fab conform instrucțiunilor producătorului pentru a realiza digestia completă a anticorpului. Fragmentele Fab rezultate au fost amestecate cu capside HPeV3 într-un raport de 60 la 1 molar și incubate timp de 30 de minute la 37 ° C în 1 x TNM pentru a permite formarea complexului virus-Fab, înainte de vitrificare.
Test de neutralizare HPeV
Neutralizarea izolatului HPeV3 152037 prin AT12-015 a fost testată prin infectarea celulelor Vero cu viruși pre-incubați (100 unități TCID50) cu cantități variabile de anticorp (0,03-7,5 µg ml -1) la 37 ° C timp de 1 oră. Celulele infectate au fost monitorizate pentru apariția CPE la fiecare 24 de ore timp de 7 zile. În ziua 7, ARN a fost extras din supernatant folosind un kit de izolare a acidului nucleic total cu instrumentul MagnaPure LC (Roche Diagnostics), transcrierea inversă și ADNc (ADN complementar) au fost utilizate pentru a estima numărul de copii virale prin PCR în timp real folosind un Instrument LC480 (Roche Diagnostics) 16. Capacitatea AT12-015 de a neutraliza izolatul HPeV3 152037 a fost, de asemenea, testată în monostratele de celule BGM (rinichi de maimuță verde bivol), A549 (adenocarcinom de colon uman) și Caco2 (adenocarcinom de colon uman). Mai mult, neutralizarea încrucișată prin AT12-015 împotriva HPeV1, HPeV2, HPeV4, HPeV5 și HPeV6 a fost, de asemenea, testată în celulele Vero. Imunofluorescența celulelor infectate cu HPeV1-6 a fost, de asemenea, verificată folosind AT12-015.
Microscopie crio-electronică și procesare de imagini.
HPeV3 purificat inactivat cu formaldehidă a fost vitrificat prin aplicarea a 3 pl din probă pe grile Quantifoil R3.5/1, uscat timp de 2 s la 92% umiditate relativă și scufundat în etan lichid folosind un Leica EM GP. Grătările au fost examinate pe un microscop electronic cu transmisie FEI Titan Krios corectat pentru Cs la 300 keV. Imaginile au fost înregistrate pe un detector Falcon II în condiții de doză mică, cu o mărire nominală de 59.000 × cu o dimensiune a eșantionului de 1,14 Å pe pixel. Șapte cadre pe imagine au fost colectate în modul de numărare utilizând software-ul FEI EPU automatizat de achiziție a unei particule. Rame de film au fost inițial aliniate între ele folosind software-ul motioncorr 17 înainte de post-procesare.
Pentru reconstrucția asimetrică, orientările celor mai bune 41.845 particule au fost rafinate pentru încă 16 runde folosind opțiunea de rafinare automată tridimensională din Relion fără a impune nicio simetrie. Rafinarea finală între două seturi de date independente a dat o rezoluție de 10,36 Å pe baza criteriului FSC 0.143.
Alicote ale complexelor HPeV3-Fab AT12-015 au fost vitrificate pe o peliculă de carbon ultra-fină descărcată termic conținând grile de cupru și carbon (TED PELLA) folosind o ghilotină auto-fabricată. Grătările au fost examinate pe un microscop electronic cu transmisie FEI Tecnai F20 la 200 keV folosind un criostat Gatan 626. Imaginile au fost înregistrate pe filme Kodak SO-163 în condiții de doză mică cu o mărire nominală de 50.000 ×. Filmele au fost digitalizate pe un scaner Zeiss (Photoscan) la o dimensiune a treptei de 7 µm, oferind o dimensiune a pixelilor de 1,4 Å pe pixel. Funcția de transfer al contrastului pentru fiecare micrografie a fost estimată așa cum s-a descris mai sus. Particulele au fost colectate în micrografii la intervale de 4 × folosind programul RobEM 21 cu o dimensiune a cutiei de 101 pixeli. Un model de pornire aleatorie a fost generat din 150 de particule folosind AUTO3DEM v 4.05.1 (ref. 21). Setul complet de date a fost împărțit în două, iar orientările și originile au fost determinate din reconstrucții independente generate de fiecare set. Rezoluția finală a 15 Å din 564 particule a fost obținută pe baza criteriului de 0,143 FSC.
Construcția modelului
Structurile celor trei proteine de capsidă HPeV3 au fost prezise prin modelarea comparativă a mai multor șabloane folosind serverul I-TASSER 25. Structurile șablon pentru VP0 au inclus un șablon definit de utilizator care conține o structură fuzionată de enterovirus uman VP4 (ID PDB: 3vbf) N terminal pentru a spăla enterovirusul uman VP2 (ID PDB: 3vbo) 26, precum și boala aftoasă. virus (PDB ID: 1qqp) 27, poliovirus 1 (PDB ID: 1pov) 28, enterovirus bovin (PDB ID: 1bev) 29, echovirus 1 (PDB ID: 1ev1) (ref. 30) și hepatită A (PDB ID: 4qpg ) 31. Scorul C pentru cel mai bun model a fost –1.03.
Pentru VP3, a fost utilizat un șablon gol furnizat de utilizator enterovirus uman (PDB ID: 3vbo) 26, pe lângă următoarele șabloane selectate de program: coxsackievirus A9 (PDB ID: 1d4m) 32, echovirus 1 (PDB ID: 1d4m) . PDB: 1ev1) (ref. 30), enterovirus uman (PDB ID: 3vbf) 26, enterovirus uman (PDB ID: 3vbh) 26, coxsackievirus uman A16 (PDB ID: 4jgy) 7. Scorul C pentru cel mai bun model a fost –0,61.
Pentru VP1, enterovirusul uman gol (ID PDB: 3vbo) 26 a fost furnizat ca șablon extern, în plus față de următoarele șabloane selectate de program: enterovirus uman 71 (ID PDB: 3vbh) 26, enterovirus uman 71 (ID PDB: 3zfe) 33, coxsackievirus uman A16 (PDB ID: 4jgy) 7, enterovirus uman 71 (PDB ID: 4cdq) 34, coxsackievirus A 9 (PDB ID: 1d4m) 32, rinovirus uman 14 (PDB ID: 1ncq) 35. Scorul C pentru cel mai bun model a fost –0,75.
Modelele de omologie au fost montate rigid pe harta HPeV3 folosind funcția „fit to map” din UCSF-Chimera 36. Folosind funcția „zonare” în UCSF-Chimera 36, harta capsidelor HPeV3 a fost împărțită într-o unitate asimetrică cu o rază de 6 Å folosind modelul VP0-VP3-VP1 cu potrivire rigidă. Modelul VP0-VP3-VP1 s-a încadrat flexibil în unitatea asimetrică folosind comparația rezultatelor a două programe diferite de ajustare flexibilă iMODfit 37 și FlexEM 38 pentru a ajunge la o potrivire consensuală 39. Ambele programe de ajustare flexibile au fost utilizate cu setările implicite. Modelele au fost apoi rafinate manual în Coot 40 prin trunchierea modelelor în care densitatea nu era evidentă, îmbunătățirea potrivirilor coloanei vertebrale și ajustarea lanțurilor laterale grele de reziduuri de fenilalanină, tirozină, triptofan și arginină acolo unde erau vizibile. Modelele au fost rafinate în continuare prin rafinarea spațiului real din Phenix 41. Toate vizualizările au fost efectuate în UCSF-Chimera 36 .
O singură copie a regiunii structurate cu ARN a fost extrasă folosind „Bulk Eraser” în UCSF-Chimera 36. Volumul extras poate conține în jur de 30 de nucleotide. O regiune de 30 nucleotide (7181-7210) la capătul 3 'netradus al genomului izolat HPeV3 152037 (GenBank ID: GQ183026) a fost pliată pe serverul web RNAfold 42 și foldul a fost modelat în RNAComposer 43. Acest model de ARN a fost adaptat la volumul extras folosind opțiunea „Potrivire pe hartă” din UCSF-Chimera 36 .
Amprenta AT12-015 a fost estimată prin suprapunerea modelului atomic HPeV3 pe reconstrucția complexului HPeV3-Fab AT12-015 și generarea foii de parcurs pentru modelul atomic cu amprenta fabulată ca linii de contur în RIVEM 44. Toate reziduurile de aminoacizi expuse la suprafață din această amprentă au fost luate ca epitopi pentru Fab.
informatii suplimentare
Coduri de acces: Hărțile de densitate ale HPeV3 icosaedric-simetric, HPeV3 asimetric și HPeV3-Fab AT12-015 au fost depuse în Banca de date cu microscopie electronică sub codurile de acces EMD-3137, EMD-3322 și EMD-3138, respectiv. Modelele ajustate pentru HPeV3 au fost depuse în banca de date cu proteine din Europa sub ID-ul PDB: 5APM. Toate datele brute colectate pentru reconstrucția HPeV3 sunt disponibile prin fișierul de imagine pilot de microscopie electronică cu codul de acces EMPIAR-10033.
Cum se citează acest articol: Shakeel, S. și colab. Mai multe interacțiuni stabilizatoare de capsidă au fost dezvăluite într-o structură de înaltă rezoluție a unui picornavirus emergent care provoacă sepsis neonatal. Nat. Comun. 7: 11387 doi: 10.1038/ncomms11387 (2016).
- Papaya, un fruct cu multiple beneficii pentru sănătate pe care ar trebui să îl includeți în dieta zilnică -
- Proprietățile multiple și benefice ale materialului seminal Placeres S Moda EL PAÍS
- Pentru o persoană înaltă de 5 picioare și 6 inci, cântărind 95 kg, care este aportul ideal de calorii?
- Femeile care doresc să slăbească ar trebui să ia o dietă bogată în proteine lactate -
- Nouă fructe cu multiple beneficii pentru piele